概要
近年、生物モニタリングにおいて、環境中に残存するDNA(環境DNA)を分析し、そこに存在する生物を網羅的に検出する「環境DNA分析」の利用が広がっています。しかし、これまでの環境DNA研究はほとんどの場合、ある種や系統がいる・いない、といった情報を引き出すのにとどまっていました。
今回、潮雅之 京都大学白眉センター特定准教授(現:香港科技大学助理教授)、益田玲爾 フィールド科学教育研究センター教授、笹野祥愛 農学研究科博士課程学生(現:水産研究・教育機構研究員)、宮正樹 千葉県立中央博物館主任研究員、長田穣 東北大学大学院生命科学研究科助教らの研究グループは、千葉県房総半島沿岸から得た魚類環境DNAの高頻度時系列データを解析することで、魚種間の関係性を検出できることを示しました。
本研究で示された環境DNA時系列データから生物間相互作用を検出する枠組みは、これまで困難であった「野外環境下での生物間相互作用の網羅的モニタリング」を可能にします。生態系の動態には生物間相互作用が大きな影響を与えています(例えば、アイゴが海藻を食べ尽くし、隠れ家を失った小魚たちが姿を消す、など)。そのため、今回の研究成果は、より正確な生態系の状態把握や近未来予測に繋がると期待されます。
本研究成果は、2023年7月11日に、国際学術誌「eLife」にVersion of recordとしてオンライン掲載されました。
今回、潮雅之 京都大学白眉センター特定准教授(現:香港科技大学助理教授)、益田玲爾 フィールド科学教育研究センター教授、笹野祥愛 農学研究科博士課程学生(現:水産研究・教育機構研究員)、宮正樹 千葉県立中央博物館主任研究員、長田穣 東北大学大学院生命科学研究科助教らの研究グループは、千葉県房総半島沿岸から得た魚類環境DNAの高頻度時系列データを解析することで、魚種間の関係性を検出できることを示しました。
本研究で示された環境DNA時系列データから生物間相互作用を検出する枠組みは、これまで困難であった「野外環境下での生物間相互作用の網羅的モニタリング」を可能にします。生態系の動態には生物間相互作用が大きな影響を与えています(例えば、アイゴが海藻を食べ尽くし、隠れ家を失った小魚たちが姿を消す、など)。そのため、今回の研究成果は、より正確な生態系の状態把握や近未来予測に繋がると期待されます。
本研究成果は、2023年7月11日に、国際学術誌「eLife」にVersion of recordとしてオンライン掲載されました。
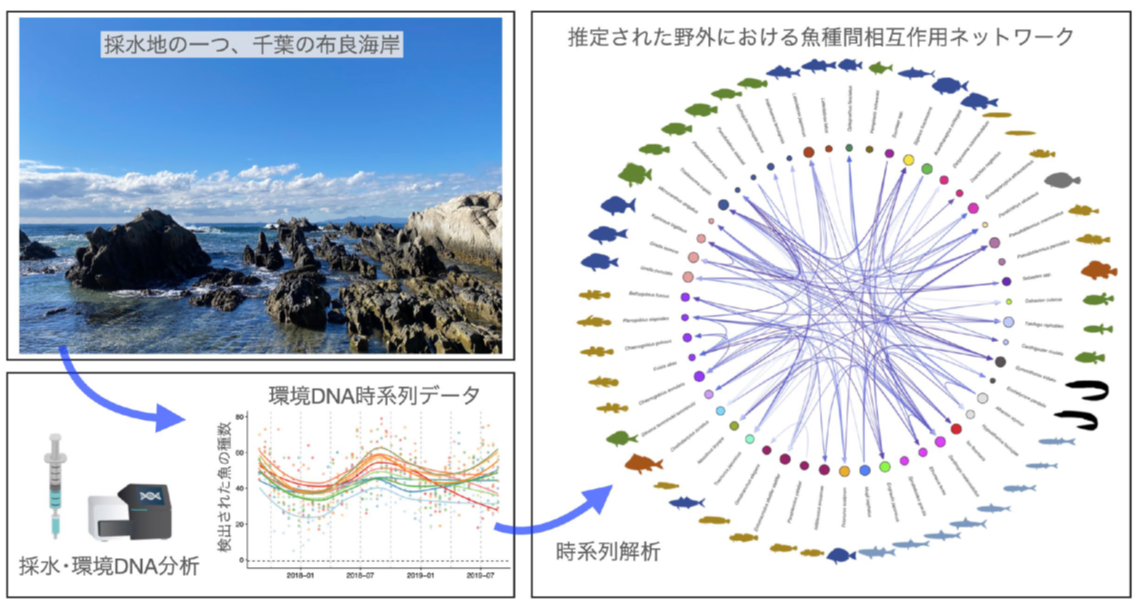
環境 DNA のサンプリングが行われた11サイトの一つ(左上;千葉の布良海岸)。得られた環境 DNA サンプルはイルミナ社の DNA シーケンサーにより解析され、環境 DNA 時系列データとなった(左下)。このデータを最先端の時系列解析法で解析し、魚種間の関係性を推定した(右図)。
詳細な説明
1.背景
生態系の状態を正確に把握することは、生態系の保全に貢献し、ひいては我々人類の生存にも貢献します。しかし、生態系の状態把握(例えば系の中にどんな生物がどのくらい生育しているのか)は簡単ではありません。生物は種や生態が多様であり、通常はそれぞれの分類群(魚や昆虫など)の生態を熟知した専門家によってそれらのモニタリングが行われてきました。近年、野外の生物モニタリングにおいて、環境中に残存する DNA、環境 DNA(注1)、を利用する方法が広がっています。生物は環境中に自身の組織由来のDNA を放出しますが、それらは環境にしばらく残留するため、環境 DNA を分析することで、環境中にいる生物を網羅的に検出することが可能になります。この「環境 DNA 分析」は、魚の検出に特によく利用され、これまで様々な魚種の網羅的検出や個体群の遺伝的構造の推定などの研究が行われてきました。
これまでの環境 DNA 研究はほとんどの場合、ある種や系統がいる・いない、といった情報を引き出すのにとどまっていました。生物種の網羅的検出は生態系の状態把握にとって重要ですが、より詳細な系の状態把握のためには、それぞれの生物種が互いにどのような関係性を持っているか、すなわち生物種間相互作用(注2)の検出・定量とその変動要因を理解する必要があります。しかし、これまで野外において生物種間相互作用が網羅的に定量された例は限られていました。あり得る相互作用の数は種数の2乗に比例するため膨大であり、かつ相互作用そのものを観測するのは容易ではないためです。特に、高い多様性と活動性のため、定量的かつ高頻度の観測が難しい沿岸生態系の魚類群集に対して、このような研究が行われた例はありませんでした。
本研究では、千葉県房総半島沿岸の 11 地点を調査地として設定し、2 週間に 1 度の採水調査を 2 年間継続しました。得たサンプルから環境 DNA を抽出し、魚類群集を環境 DNA メタバーコーディング(多種同時並列検出)(注3)という手法で分析し、沿岸生態系の魚類群集の時間動態を詳細に調べました。さらに得られた多魚種・多地点・高頻度の環境 DNA 時系列データを最先端の時系列解析法で解析することで、魚種間に働く相互作用強度を検出・定量することに挑戦しました。
生態系の状態を正確に把握することは、生態系の保全に貢献し、ひいては我々人類の生存にも貢献します。しかし、生態系の状態把握(例えば系の中にどんな生物がどのくらい生育しているのか)は簡単ではありません。生物は種や生態が多様であり、通常はそれぞれの分類群(魚や昆虫など)の生態を熟知した専門家によってそれらのモニタリングが行われてきました。近年、野外の生物モニタリングにおいて、環境中に残存する DNA、環境 DNA(注1)、を利用する方法が広がっています。生物は環境中に自身の組織由来のDNA を放出しますが、それらは環境にしばらく残留するため、環境 DNA を分析することで、環境中にいる生物を網羅的に検出することが可能になります。この「環境 DNA 分析」は、魚の検出に特によく利用され、これまで様々な魚種の網羅的検出や個体群の遺伝的構造の推定などの研究が行われてきました。
これまでの環境 DNA 研究はほとんどの場合、ある種や系統がいる・いない、といった情報を引き出すのにとどまっていました。生物種の網羅的検出は生態系の状態把握にとって重要ですが、より詳細な系の状態把握のためには、それぞれの生物種が互いにどのような関係性を持っているか、すなわち生物種間相互作用(注2)の検出・定量とその変動要因を理解する必要があります。しかし、これまで野外において生物種間相互作用が網羅的に定量された例は限られていました。あり得る相互作用の数は種数の2乗に比例するため膨大であり、かつ相互作用そのものを観測するのは容易ではないためです。特に、高い多様性と活動性のため、定量的かつ高頻度の観測が難しい沿岸生態系の魚類群集に対して、このような研究が行われた例はありませんでした。
本研究では、千葉県房総半島沿岸の 11 地点を調査地として設定し、2 週間に 1 度の採水調査を 2 年間継続しました。得たサンプルから環境 DNA を抽出し、魚類群集を環境 DNA メタバーコーディング(多種同時並列検出)(注3)という手法で分析し、沿岸生態系の魚類群集の時間動態を詳細に調べました。さらに得られた多魚種・多地点・高頻度の環境 DNA 時系列データを最先端の時系列解析法で解析することで、魚種間に働く相互作用強度を検出・定量することに挑戦しました。
2.研究手法・成果
本研究ではまず、千葉県房総半島沿岸にほぼ等間隔で 11 地点の調査地を設定しました。2週間に1度調査地を訪れ、1L の水を採水・フィルターで現場ろ過し、実験室に持ち帰り、フィルターから環境 DNA を抽出しました。調査は2年間継続され、合計 550 の環境 DNA サンプルを得ました。その後、研究グループのメンバーが過去に開発した MiFish プライマー(注4)を用いて環境 DNA サンプル中の魚類 DNA を増幅し、イルミナ社のシーケンサーを用いて配列を解析しました。また、ほとんどのサンプルから検出されたクロダイの環境 DNA を定量し、内部標準として扱うことで各魚種の環境 DNA 量の推定も行いました。
その結果、解析した 550 サンプルから合計 856 種の魚の DNA を検出しました。この種数は文献調査や博物館のサンプルから得られる 948 種という種数に近いものでした。また、種数・環境 DNA 量(魚の量の指標)はともに夏に高く、冬に低い、という傾向が見て取れ、これも既存の情報と一致するものでした。
魚種間の野外環境における相互作用強度を推定するために、優占的であった 50 魚種の環境 DNA 時系列データに近年開発された2つの時系列解析法(注5)を適用しました。その結果、行動観察から得られる結果と矛盾しない、魚種間の相互作用が検出されました。さらに、これらの相互作用が水温から強い影響を受けていることが分かりました。
本研究ではまず、千葉県房総半島沿岸にほぼ等間隔で 11 地点の調査地を設定しました。2週間に1度調査地を訪れ、1L の水を採水・フィルターで現場ろ過し、実験室に持ち帰り、フィルターから環境 DNA を抽出しました。調査は2年間継続され、合計 550 の環境 DNA サンプルを得ました。その後、研究グループのメンバーが過去に開発した MiFish プライマー(注4)を用いて環境 DNA サンプル中の魚類 DNA を増幅し、イルミナ社のシーケンサーを用いて配列を解析しました。また、ほとんどのサンプルから検出されたクロダイの環境 DNA を定量し、内部標準として扱うことで各魚種の環境 DNA 量の推定も行いました。
その結果、解析した 550 サンプルから合計 856 種の魚の DNA を検出しました。この種数は文献調査や博物館のサンプルから得られる 948 種という種数に近いものでした。また、種数・環境 DNA 量(魚の量の指標)はともに夏に高く、冬に低い、という傾向が見て取れ、これも既存の情報と一致するものでした。
魚種間の野外環境における相互作用強度を推定するために、優占的であった 50 魚種の環境 DNA 時系列データに近年開発された2つの時系列解析法(注5)を適用しました。その結果、行動観察から得られる結果と矛盾しない、魚種間の相互作用が検出されました。さらに、これらの相互作用が水温から強い影響を受けていることが分かりました。
3.波及効果、今後の予定
本研究では、近年発展が著しい環境 DNA 分析に最先端の時系列解析法を適用するため、注意深い調査デザインや慎重なサンプル分析が必要でした。忍耐強くそれらを実行することで、野外環境において多種多様な魚の種間関係がどうなっており、その強さが何によって決まるのか、その一端を明らかにすることが出来ました。異分野の最先端手法と融合させることで、環境 DNA 分析で「何がいるか」「どのくらいいるか」だけでなく、「何と何がどのくらいの強さで関係しているか」まで推定できることを示しました。この研究の枠組みは、他の生物分類群にも応用可能です。将来的には、例えば水産重要種に病気を及ぼしうる生物の特定などにも貢献できるかもしれません。また、生態系の隠れたキープレイヤーを検出できるかもしれません。
一方で、環境 DNA 分析そのものの更なる改良、例えば生物量推定能力の向上、が望まれます。また、DNA 配列解読技術の向上により、今後さらに大規模な環境 DNA データセットが次々と生み出されるでしょう。したがって、それらを解析するための高精度・高速なデータ解析法の開発も望まれます。環境 DNA 分析技術とデータ解析技術それぞれの向上、さらにそれらの協働により、現場での「採水」からさらに高精度・高効率な生態系モニタリングが可能になるでしょう。
本研究では、近年発展が著しい環境 DNA 分析に最先端の時系列解析法を適用するため、注意深い調査デザインや慎重なサンプル分析が必要でした。忍耐強くそれらを実行することで、野外環境において多種多様な魚の種間関係がどうなっており、その強さが何によって決まるのか、その一端を明らかにすることが出来ました。異分野の最先端手法と融合させることで、環境 DNA 分析で「何がいるか」「どのくらいいるか」だけでなく、「何と何がどのくらいの強さで関係しているか」まで推定できることを示しました。この研究の枠組みは、他の生物分類群にも応用可能です。将来的には、例えば水産重要種に病気を及ぼしうる生物の特定などにも貢献できるかもしれません。また、生態系の隠れたキープレイヤーを検出できるかもしれません。
一方で、環境 DNA 分析そのものの更なる改良、例えば生物量推定能力の向上、が望まれます。また、DNA 配列解読技術の向上により、今後さらに大規模な環境 DNA データセットが次々と生み出されるでしょう。したがって、それらを解析するための高精度・高速なデータ解析法の開発も望まれます。環境 DNA 分析技術とデータ解析技術それぞれの向上、さらにそれらの協働により、現場での「採水」からさらに高精度・高効率な生態系モニタリングが可能になるでしょう。
4.研究プロジェクトについて
本プロジェクトは下記の支援を受けて実施しました:京都大学白眉プロジェクト、科研費基盤 B(20H03323) (潮)、香港科技大学スタートアップファンド、科研費基盤 B (19H03291, 22H02691) (宮),
MEXT OGAP プロジェクト (JPMXD0618068274) (宮)
本プロジェクトは下記の支援を受けて実施しました:京都大学白眉プロジェクト、科研費基盤 B(20H03323) (潮)、香港科技大学スタートアップファンド、科研費基盤 B (19H03291, 22H02691) (宮),
MEXT OGAP プロジェクト (JPMXD0618068274) (宮)
<用語解説>
注1)環境 DNA
環境中、たとえば水や土の中に残存する生物由来の DNA の総称。環境 DNA の配列を分析することで、その環境中に生息している生物を検出できる。従来は微生物の検出に用いられることが多かったが、近年、魚類など水生生物の分布調査への利用が急速に拡大している。
注1)環境 DNA
環境中、たとえば水や土の中に残存する生物由来の DNA の総称。環境 DNA の配列を分析することで、その環境中に生息している生物を検出できる。従来は微生物の検出に用いられることが多かったが、近年、魚類など水生生物の分布調査への利用が急速に拡大している。
注 2)生物種間相互作用
例えば「食う−食われる」「同じ資源を巡って競争する」「共生する」といった生物種同士の関係性を指す。多くの理論研究や実験環境下での研究によって、生物種間相互作用が群集全体の動態(個体数変動や共存可能な種数など)に影響を与えることが示されているが、野外環境下で多種を対象として生物種間相互作用を網羅的に調べ、それらと群集動態の関係を調べた研究は多くない。
例えば「食う−食われる」「同じ資源を巡って競争する」「共生する」といった生物種同士の関係性を指す。多くの理論研究や実験環境下での研究によって、生物種間相互作用が群集全体の動態(個体数変動や共存可能な種数など)に影響を与えることが示されているが、野外環境下で多種を対象として生物種間相互作用を網羅的に調べ、それらと群集動態の関係を調べた研究は多くない。
注 3)メタバーコーディング(多種同時並列検出)
解析対象の生物分類群(例えば、細菌)が共通して持っている DNA 領域を増幅し、網羅的に配列を解読することで、多種検出を同時に行う DNA 解析技術。本研究では特に、ほとんどのサンプルから検出されるクロダイの環境 DNA 濃度を定量することで、単位 DNA 量あたり生成される配列数を推定し、他の魚種の DNA 量も網羅的に推定した(定量的環境 DNA メタバーコーディング)。
解析対象の生物分類群(例えば、細菌)が共通して持っている DNA 領域を増幅し、網羅的に配列を解読することで、多種検出を同時に行う DNA 解析技術。本研究では特に、ほとんどのサンプルから検出されるクロダイの環境 DNA 濃度を定量することで、単位 DNA 量あたり生成される配列数を推定し、他の魚種の DNA 量も網羅的に推定した(定量的環境 DNA メタバーコーディング)。
注 4)MiFish プライマー
メタバーコーディングを用いた研究において、魚類の環境 DNA を増幅するために用いられる 21-27 塩基の短い DNA 配列。非常に多くの魚の DNA を増幅し、かつ増幅配列を用いて判別できる魚種も多いため、魚類環境 DNA メタバーコーディングにおいて、もっとも一般的に使用されているプライマーの一つとなっている。
メタバーコーディングを用いた研究において、魚類の環境 DNA を増幅するために用いられる 21-27 塩基の短い DNA 配列。非常に多くの魚の DNA を増幅し、かつ増幅配列を用いて判別できる魚種も多いため、魚類環境 DNA メタバーコーディングにおいて、もっとも一般的に使用されているプライマーの一つとなっている。
注 5)近年開発された2つの時系列解析法
近年、時系列解析に基づいて変数間の因果を検出するための手法が活発に開発されているが、本研究では情報理論に基づいた Unified Information-theoretic Causality (UIC)と力学系理論に基づく Multiview distance regularized S-map (MDR S-map)という手法を用いた。UIC により魚種間の因果関係を検出し、MDR Smapにより因果を持つ魚種ペアに対して相互作用強度を定量した。
近年、時系列解析に基づいて変数間の因果を検出するための手法が活発に開発されているが、本研究では情報理論に基づいた Unified Information-theoretic Causality (UIC)と力学系理論に基づく Multiview distance regularized S-map (MDR S-map)という手法を用いた。UIC により魚種間の因果関係を検出し、MDR Smapにより因果を持つ魚種ペアに対して相互作用強度を定量した。
<論文タイトルと著者>
タイトル:Temperature sensitivity of the interspecific interaction strength of coastal marine fish communities(沿岸魚類群集の種間相互作用強度の温度感受性)
著 者:Masayuki Ushio, Tetsuya Sado, Takehiko Fukuchi, Sachia Sasano, Reiji Masuda, Yutaka
Osada, Masaki Miya
掲 載 誌:eLife
DOI: 10.7554/eLife.85795
関連リンク
問い合わせ先
(報道に関すること)
東北大学大学院生命科学研究科 広報室
電話番号:022-217-6193
Eメール:lifsci-pr@grp.tohoku.ac.jp

