発表のポイント
- 日本各地で採取された136系統のミヤコグサのゲノムを解読し、遺伝的な関係性を明らかにした。
- ゲノム配列の比較から、日本のミヤコグサは3つのグループに分けられることを発見した。
- 東北大学の鹿島台圃場での栽培実験の結果から、越冬性と開花調節に関連する遺伝子が関東・東北・北海道に自生する系統の地域適応の鍵であったことを明らかにした。
- 本研究で日本の環境への適応に機能した遺伝子が具体的に同定されたことにより、環境適応の分子メカニズムの解明に向けた研究が加速されることが期待される。
概要
貝原益軒が編纂した「大和本草」にも取り上げられているミヤコグサは、日本全土に自生するマメ科の野草であり、南北に長く多様な日本の気候環境に適応してきました。東北大学大学院生命科学研究科の佐藤修正准教授らのグループは、デンマーク・オーフス大のStig Andersen准教授、Mikkel Schierup教授、宮崎大学農学部の明石 良教授をはじめとする国際研究グループと共同で、日本各地に自生するマメ科植物のミヤコグサが自生地の環境に適応してきた過程を明らかにしました。日本各地から採取した136系統のミヤコグサのゲノム解析と、東北大学の圃場での栽培実験により、ミヤコグサが約2万年前に九州に定着し、その後日本全土に分布域を広げたことを見出しました。更に、北日本地域への適応には、越冬性と開花調節に関わる遺伝子が鍵になったことを明らかにしました。
本研究成果は、1月14日付けでNature Communications誌に掲載されました。
本研究は、AMED ナショナルバイオリソースプロジェクト・ゲノム情報等整備プログラムおよびJST戦略的創造研究推進事業 CRESTの支援を受けて行われました。
詳細な説明
ミヤコグサは日本全土に自生するマメ科の野草であり、歳時記にも取り上げられている身近な植物です。このミヤコグサはマメ科のモデル植物として微生物共生の研究を中心に海外を含む多くの研究者に使われてきた経緯があり、かずさDNA研究所によるゲノム配列情報解読やナショナルバイオリソースプロジェクト(NBRP)による研究リソースの整備も行われています。そのような研究リソースの一つに、日本各地で採取されたミヤコグサ野生系統があります。今回の研究は、このミヤコグサ野生系統の情報基盤整備の一環として行ったもので、南北に長く多様な日本の気候環境に適応してきたミヤコグサの歴史をゲノム情報から紐解くことを試みました。
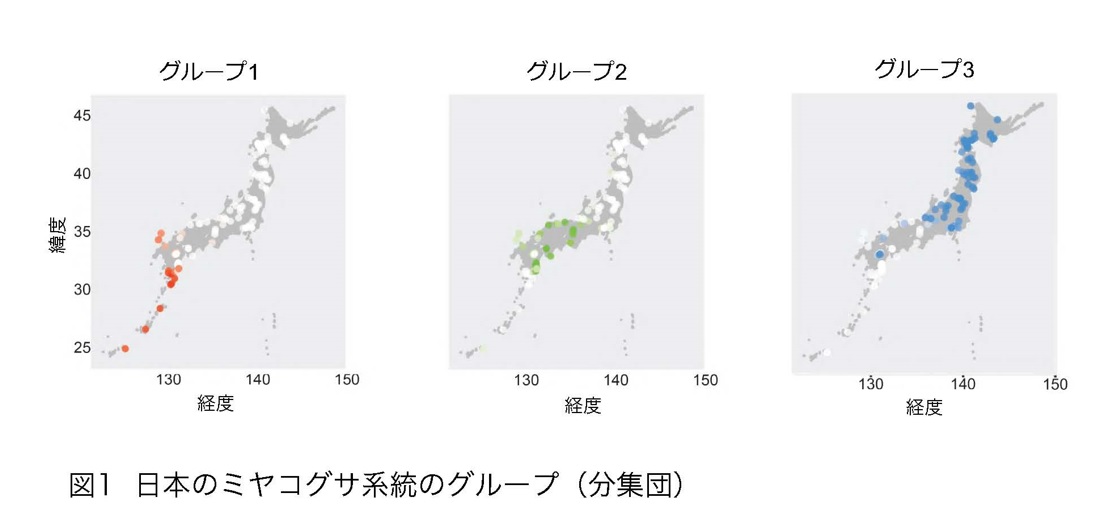
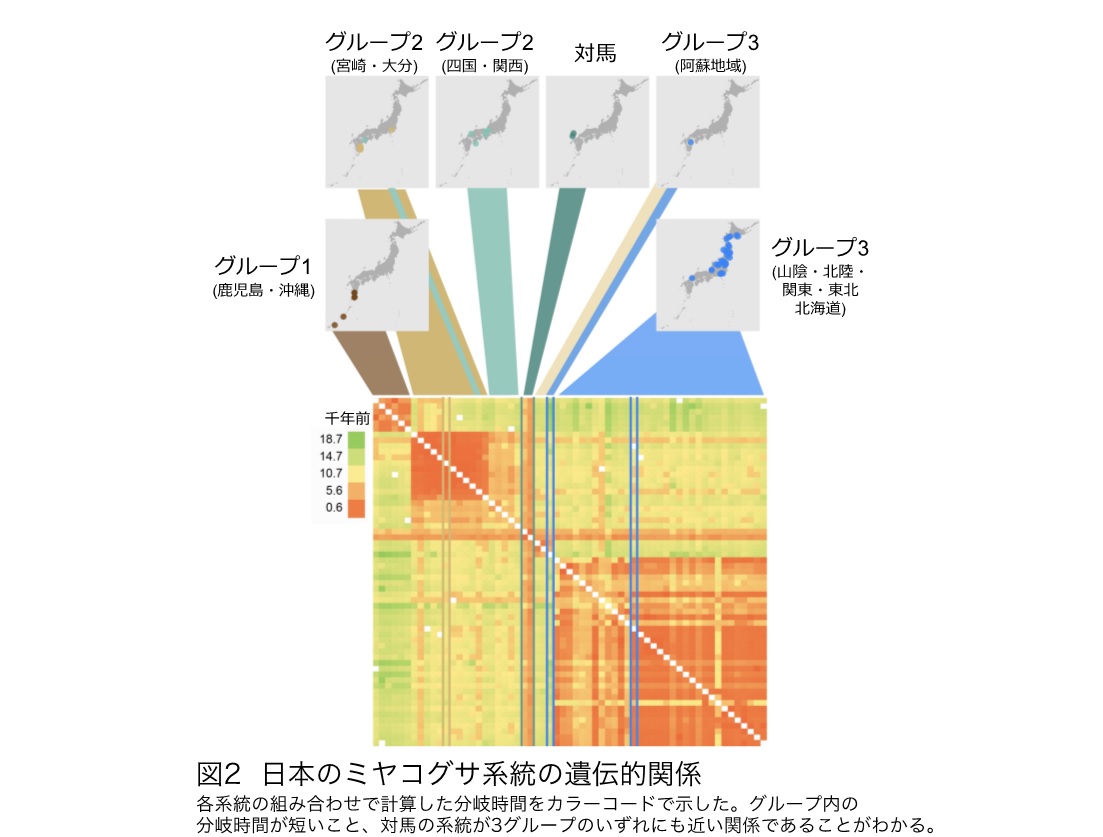
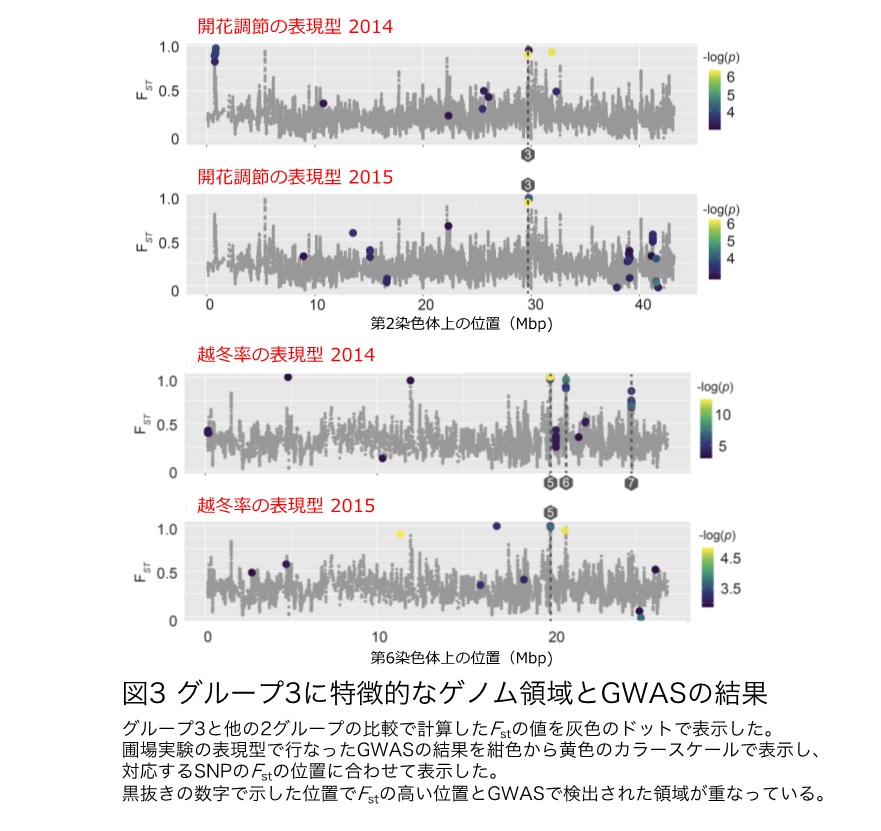
まず、NBRPで収集されているミヤコグサ系統のうち136系統のゲノムを解読し、比較解析を行いました。その結果、日本のミヤコグサは鹿児島・沖縄に自生する系統(グループ1)、九州東岸・四国・関西に自生する系統(グループ2)、九州の阿蘇地域・山陰・北陸・関東・東北・北海道に自生する系統(グループ3)の3グループ(分集団)に分けられることを見出しました(図1)。系統間の塩基の置換率を基に計算した結果、これらのグループが形成された時期は1万〜1万8千年前と推定され、長崎県の対馬に自生する系統がこれら3グループの祖先的な性質を持つことを見出しました(図2)。また、同定されたグループ間での比較解析を行った結果、グループ3で特徴的に選抜されているゲノム領域(Fst*1が高い領域)が検出され、グループ3が高緯度地域に適応する過程で選択を受けた遺伝子の候補を見つけることができました(図3)。
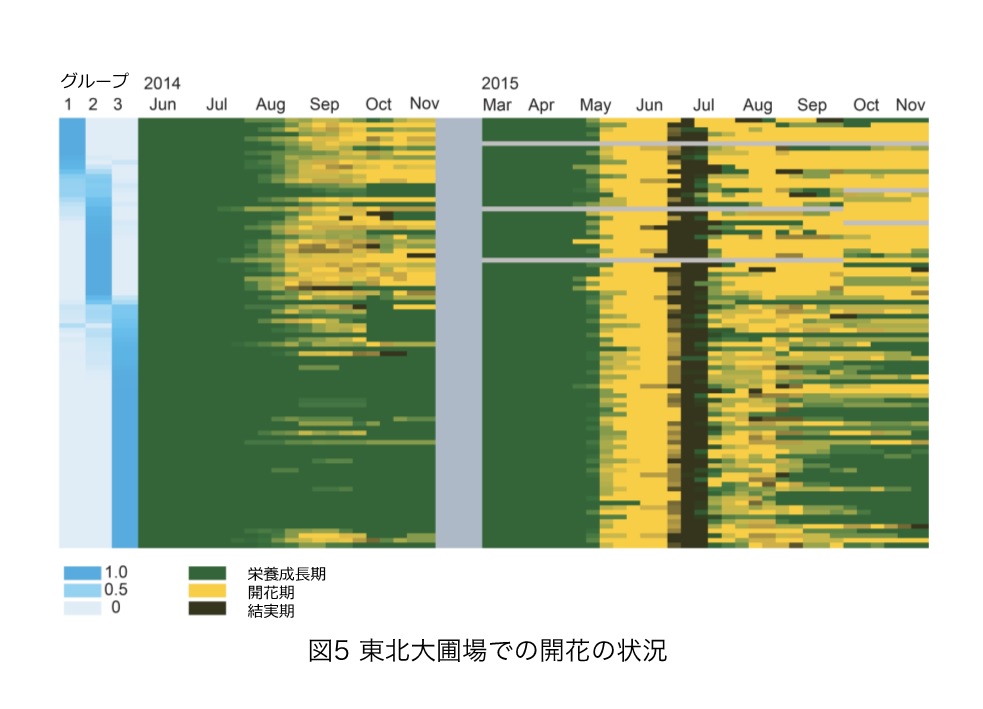
一方で、ゲノム解読を行なった系統を東北大学の鹿島台圃場(宮城県大崎市)で栽培し、生育や開花、塩ストレスに対する反応など、さまざまな表現型について調査しました。東北大圃場でミヤコグサを栽培すると、冬の間は地上部は枯れますが、ミヤコグサは多年生植物であるため、春には新芽を出し5月から6月にかけて一斉に花を咲かせます(図4, 5)。この春に新芽を出した個体の割合を越冬率として評価すると、グループ3に属する高緯度地域の系統はほぼ全ての個体が越冬できたのに対し、グループ1や2に属する低緯度地域の系統は越冬できない個体が多く検出されました(図4)。逆に、宮崎大学の圃場で栽培すると、グループ1や2に属する低緯度地域の系統は冬枯れすることなく2月には新芽を出すのに対して、グループ3に属する高緯度地域の系統は夏以降の生育が悪く越冬できない個体が多く検出されました(図4)。また、東北大圃場での栽培では、グループ1や2に属する系統は冬枯れが始まる12月初旬まで開花している個体が見られるのに対し、グループ3に属する系統は9月以降に開花を抑制する傾向が見られました(図5)。得られた表現型に関与する遺伝子を探索する目的でゲノムワイド関連解析(GWAS)*2を行った結果、越冬性に関する表現型と開花の調節に関する表現型について、検出されたSNP*3が、グループ間のゲノム比較においてグループ3で特徴的に選抜されているゲノム領域と一致することが明らかになりました(図3)。表現型を考慮しない集団間の遺伝的な解析で見出された遺伝子と、大規模な圃場実験での表現型を用いた集団構造を考慮したGWASの結果が一致したことにより、越冬性に関する遺伝子と開花の調節に関する遺伝子が地域適応に関与したことが強く示唆される結果になりました。
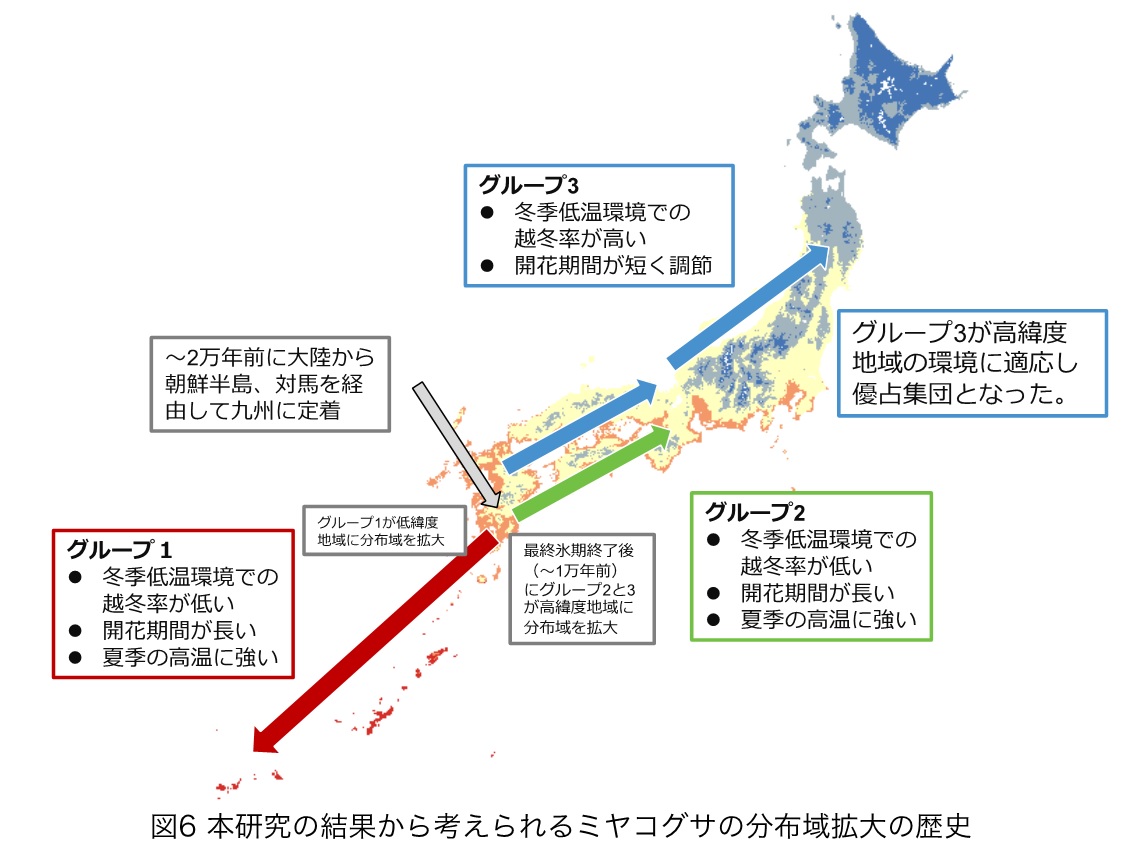
本研究で得られた結果を総合すると、原産地の中央アジアから東に分布域を拡大したミヤコグサは朝鮮半島から、対馬を経由して九州に定着し、そこで分集団を形成し、最終氷期が終わると、グループ2とグループ3が高緯度地域に分布域を拡大し、そのうちより高緯度の環境に適応したグループ3が関東・東北・北海道の優占系統になったと考えられます(図6)。本研究で日本の環境への適応に機能した遺伝子が具体的に同定されたことにより、整備が進んでいるミヤコグサの研究リソースを活用して、環境適応の分子メカニズムの解明に向けた研究が加速されることが期待されます。
まず、NBRPで収集されているミヤコグサ系統のうち136系統のゲノムを解読し、比較解析を行いました。その結果、日本のミヤコグサは鹿児島・沖縄に自生する系統(グループ1)、九州東岸・四国・関西に自生する系統(グループ2)、九州の阿蘇地域・山陰・北陸・関東・東北・北海道に自生する系統(グループ3)の3グループ(分集団)に分けられることを見出しました(図1)。系統間の塩基の置換率を基に計算した結果、これらのグループが形成された時期は1万〜1万8千年前と推定され、長崎県の対馬に自生する系統がこれら3グループの祖先的な性質を持つことを見出しました(図2)。また、同定されたグループ間での比較解析を行った結果、グループ3で特徴的に選抜されているゲノム領域(Fst*1が高い領域)が検出され、グループ3が高緯度地域に適応する過程で選択を受けた遺伝子の候補を見つけることができました(図3)。
一方で、ゲノム解読を行なった系統を東北大学の鹿島台圃場(宮城県大崎市)で栽培し、生育や開花、塩ストレスに対する反応など、さまざまな表現型について調査しました。東北大圃場でミヤコグサを栽培すると、冬の間は地上部は枯れますが、ミヤコグサは多年生植物であるため、春には新芽を出し5月から6月にかけて一斉に花を咲かせます(図4, 5)。この春に新芽を出した個体の割合を越冬率として評価すると、グループ3に属する高緯度地域の系統はほぼ全ての個体が越冬できたのに対し、グループ1や2に属する低緯度地域の系統は越冬できない個体が多く検出されました(図4)。逆に、宮崎大学の圃場で栽培すると、グループ1や2に属する低緯度地域の系統は冬枯れすることなく2月には新芽を出すのに対して、グループ3に属する高緯度地域の系統は夏以降の生育が悪く越冬できない個体が多く検出されました(図4)。また、東北大圃場での栽培では、グループ1や2に属する系統は冬枯れが始まる12月初旬まで開花している個体が見られるのに対し、グループ3に属する系統は9月以降に開花を抑制する傾向が見られました(図5)。得られた表現型に関与する遺伝子を探索する目的でゲノムワイド関連解析(GWAS)*2を行った結果、越冬性に関する表現型と開花の調節に関する表現型について、検出されたSNP*3が、グループ間のゲノム比較においてグループ3で特徴的に選抜されているゲノム領域と一致することが明らかになりました(図3)。表現型を考慮しない集団間の遺伝的な解析で見出された遺伝子と、大規模な圃場実験での表現型を用いた集団構造を考慮したGWASの結果が一致したことにより、越冬性に関する遺伝子と開花の調節に関する遺伝子が地域適応に関与したことが強く示唆される結果になりました。
本研究で得られた結果を総合すると、原産地の中央アジアから東に分布域を拡大したミヤコグサは朝鮮半島から、対馬を経由して九州に定着し、そこで分集団を形成し、最終氷期が終わると、グループ2とグループ3が高緯度地域に分布域を拡大し、そのうちより高緯度の環境に適応したグループ3が関東・東北・北海道の優占系統になったと考えられます(図6)。本研究で日本の環境への適応に機能した遺伝子が具体的に同定されたことにより、整備が進んでいるミヤコグサの研究リソースを活用して、環境適応の分子メカニズムの解明に向けた研究が加速されることが期待されます。
【用語説明】
*1 Fst:
固定指数、集団間が遺伝的にどれぐらい違うかを示す指数。0 から 1の値を取り、遺伝的な違い(分化)が大きいほど大きな値を示す。
*1 Fst:
固定指数、集団間が遺伝的にどれぐらい違うかを示す指数。0 から 1の値を取り、遺伝的な違い(分化)が大きいほど大きな値を示す。
*2 ゲノムワイド関連解析(GWAS):
遺伝子多型を用いて表現型と関連する遺伝子を見つける方法の1つ。ある表現型とSNPの遺伝子型の間で、相関があるかどうかを統計的に検定して調べる。ミヤコグサの解析の場合、ゲノム全体を網羅する20万カ所のSNPを用いて、ゲノム全体から表現型と関連する領域・遺伝子を発見する。
遺伝子多型を用いて表現型と関連する遺伝子を見つける方法の1つ。ある表現型とSNPの遺伝子型の間で、相関があるかどうかを統計的に検定して調べる。ミヤコグサの解析の場合、ゲノム全体を網羅する20万カ所のSNPを用いて、ゲノム全体から表現型と関連する領域・遺伝子を発見する。
*3 一塩基多型(SNP):
集団のゲノム配列中でみられる一塩基レベルの違い(多様性)。SNPはSingle Nucleotide Polymorphismの略。
集団のゲノム配列中でみられる一塩基レベルの違い(多様性)。SNPはSingle Nucleotide Polymorphismの略。

【論文情報】
題目:Extreme genetic signatures of local adaptation during Lotus japonicus clonization of Japan
著者:Niraj Shah, Tomomi Wakabayashi, Yasuko Kawamura, Cathrine Kiel Skovbjerg, Ming-Zhuo Wang, Yusdar Mustamin, Yoshiko Isomura, Vikas Gupta, Haojie Jin, Terry Mun, Niels Sandal, Fuyuki Azuma, Eigo Fukai, Ümit Seren, Shohei Kusakabe, Yuki Kikuchi, Shogo Nitanda, Takashi Kumaki, Mads Sønderkær, Kaare Lehmann Nielsen, Korbinian Schneeberger, Jens Stougaard, Shusei Sato*, Mikkel Heide Schierup*, and Stig Uggerhøj Andersen
著者:Niraj Shah, Tomomi Wakabayashi, Yasuko Kawamura, Cathrine Kiel Skovbjerg, Ming-Zhuo Wang, Yusdar Mustamin, Yoshiko Isomura, Vikas Gupta, Haojie Jin, Terry Mun, Niels Sandal, Fuyuki Azuma, Eigo Fukai, Ümit Seren, Shohei Kusakabe, Yuki Kikuchi, Shogo Nitanda, Takashi Kumaki, Mads Sønderkær, Kaare Lehmann Nielsen, Korbinian Schneeberger, Jens Stougaard, Shusei Sato*, Mikkel Heide Schierup*, and Stig Uggerhøj Andersen
雑誌:Nature Communications
DOI:https://doi.org/10.1038/s41467-019-14213-y
DOI:https://doi.org/10.1038/s41467-019-14213-y
【問い合わせ先】
<研究に関すること>
東北大学大学院生命科学研究科
担当:佐藤 修正(さとう しゅうせい)
電話番号:022-217-5688
E-mail:shuseis(at)ige.tohoku.ac.jp
担当:佐藤 修正(さとう しゅうせい)
電話番号:022-217-5688
E-mail:shuseis(at)ige.tohoku.ac.jp
<報道に関すること>
東北大学大学院生命科学研究科
担当:高橋 さやか (たかはし さやか)
電話番号: 022-217-6193
E-mail: lifsci-pr(at)grp.tohoku.ac.jp

