研究内容
トランスクリプトーム解析、比較ゲノム解析、分子生物学実験の融合による「虫こぶ」誘導機構の解明
「虫こぶ」とは、昆虫による摂食や産卵の影響で宿主植物の細胞や組織が過増殖や過成長して生じる、異形な器官です。92科570種の植物が宿主として確認されていて、 虫こぶの形態は多様性に富んでいます。寄生昆虫が異なれば同一宿主植物上でも異なった形態の虫こぶが形成されることから、昆虫種により異なる虫こぶ誘導因子が存在し、また宿主植物側の応答も誘導因子によって異なると考えられます。このような多様な形態を示す「虫こぶ」の分子機構はいまだ明らかになっていません。私たちはトランスクリプトーム解析、比較ゲノム解析、分子生物学的手法によりその分子機構を明らかにすることを目指して研究を行なっています。

比較ゲノム解析による高再生能力種特異的遺伝子の探索
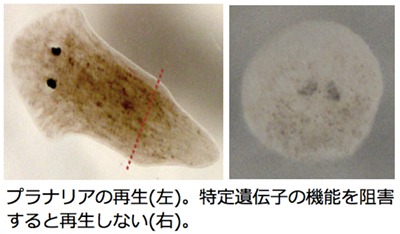
現在では多くの生物のゲノム配列が利用可能になり、今までできなかったアプローチで、特定の表現型に関わる遺伝子の探索が可能になってきました。私たちは、比較ゲノム解析により高い再生能力をもつ生物種が特異的に持つ遺伝子の探索を行なっています。再生能力は動物種間で大きく異なり、プラナリアやネマトステラのような再生能力の高い種では、切断された断片から全身を再生できる一方で、センチュウのように再生能力が低い種も存在します。種によって再生能力が異なる背景にある分子基盤や、こうした違いに繋がる進化過程はよく分かっていません。私たちは、再生能力が高い種と低い種のゲノムを比較することで、高再生能力種に特異的遺伝子を見出しました(Cao P. et al., GBE 2019)。高再生能力種であるプラナリアを対象に、この再生関連遺伝子のRNAiノックダウンを実施した結果、多くのプラナリア個体が再生せずに死亡しました。現在、この再生関連遺伝子の分子機能や再生過程における役割の解明を目指して研究を進めています。
全ゲノム重複による量的均衡遺伝子の進化
全てのゲノムが重複する特殊なイベント全ゲノム重複(WGD)が起こると、全ての遺伝子がコピーを持ちます。このとき相対的な遺伝子量は変化しないため、遺伝子量変化に脆弱な量的均衡遺伝子(DBG)も有害な影響を受けることなくコピーを作ることができます。全ゲノム重複によって生じた重複遺伝子は、全ゲノム重複による進化を提唱された大野乾博士にちなんでオオノログと呼ばれています。マウスにおいてオオノログのノックアウトを行うと致死となる傾向を発見し、オノログが遺伝子量変化に脆弱な量的均衡遺伝子であることを提案しました (Makino T. et al., Trends in Genetics 2009)。
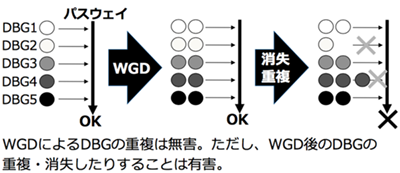
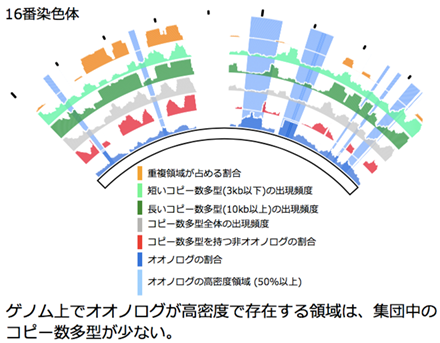
オオノログが量的均衡遺伝子であることを進化学的視点から検証するため、比較ゲノム解析により複数の動物を用いて進化過程における遺伝子の重複パターンを調査しました。その結果、オオノログが進化過程において重複しにくい遺伝子群であることを突き止めました (Makino T. et al., PNAS 2010)。さらに、オオノログの量的均衡性に着目し、ゲノム上でオオノログに隣接する遺伝子はコピー数多型がない傾向にあり、逆にオオノログから離れて存在する遺伝子の多くがコピー数多型を持つことを示しました。特に、オオノログが高密度で存在するゲノム領域では、コピー数多型が強く抑制されており、ヒトゲノム上に遺伝子重複砂漠が存在すること発見しました(Makino T. et al., Nature Communications 2013)。現在は、量的均衡遺伝子が重複しにくい性質を利用して、動物の進化過程で起きた全ゲノム重複時期の推定に取り組んでいます。
全ゲノム重複研究の医学分野への応用
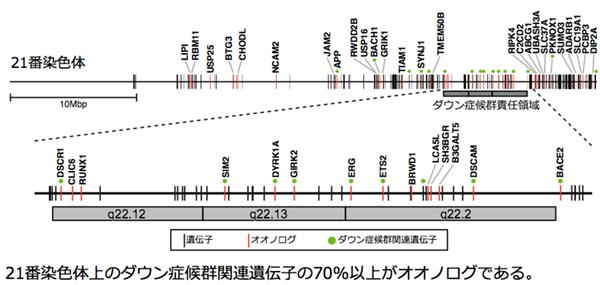
オオノログには疾患関連遺伝子が多く含まれることが分かってきました。例えば、これまでに報告されているダウン症候群関連遺伝子の75%がオオノログでした(Makino T. et al., PNAS 2010)。この他にも量的均衡性の知見を活かして症状が出にくいことから研究の進んでいなかった性染色体数異常疾患であるクラインフェルター症候群・ターナー症候群 (Pessia E., Makino T. et al., PNAS 2012)や、精神疾患 (McLysaght A., Makino T. et al., PNAS 2014)、アルツハイマー症 (Sekine M. and Makino T. MBE 2017)などの多数の疾患原因遺伝子を推定し、進化学的研究を医学へ応用する新たな手法を示してきました。オオノログ以外の遺伝子量感受性のゲノム領域にも着目し、ゲノム解析から創出された新しい疾患原因遺伝子の予測手法の確立を目指しています。
遺伝子重複研究の生態学分野への応用
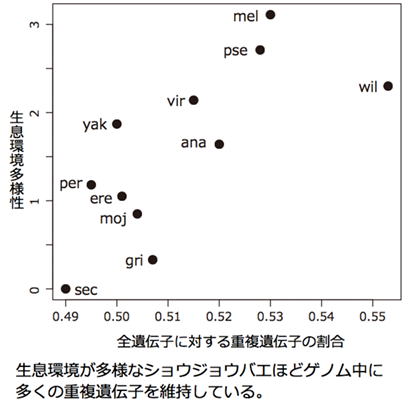
生物は様々な環境に生息していますが、生息環境の多様性は種によって異なります。生息可能な環境幅が広い種は、他種よりも環境適応能力が高いと考えられますが、生息環境の多様性を決める要因は不明です。私たちは、遺伝子が重複後に遺伝的変異を蓄積しやすく新規機能を生じやすい点に着目し、多様な環境に生息している生物種ほど多くの重複遺伝子をゲノム中に維持していると考えました。ゲノム配列が既知であるショウジョウバエ属11種のゲノム中の重複遺伝子保持率と生息環境多様性との関係を調べた結果、これらに強い正の相関があることが明らかとなりました (Makino T. and Kawata M. MBE 2012)。また、哺乳動物30種のゲノムデータを用いた解析においても同様の傾向を見出しており、ゲノム中の重複遺伝子保持率と生息環境多様性の相関に一般性があることが強く示唆されました (Tamate SC et al., MBE 2014)。これらの結果は、生物が持つ環境適応力を、ゲノム配列から評価できる可能性を示しています。現在は、本研究の応用をさらに推進し、ゲノム科学の視点で生態学分野における大きな課題である侵略種(Makino T. and Kawata M. Mol. Ecol. 2019)と絶滅危惧種(Hamabata et al. Commun. Biol. 2019)の選定を行う新しいアプローチに取り組んでいます。
家畜種・栽培種ゲノム中における有害変異蓄積
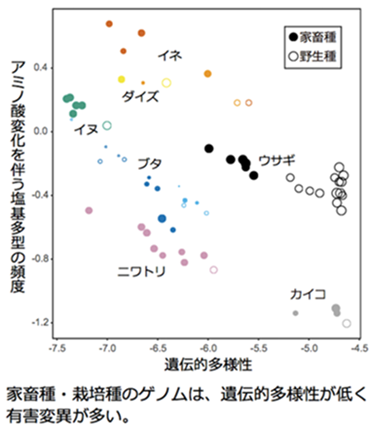
どのような生物集団においても、個体の適応度を下げる有害な遺伝的変異が存在しています。動物の家畜化や植物の栽培化は、集団ボトルネックや人為選択の影響により、ゲノム中の有害変異の割合を増加させると考えられています。しかし、こうした影響が家畜種・栽培種においてどの程度一般的な特徴と言えるのか不明でした。そこで、私たちは、家畜種5種(イヌ、ブタ、ウサギ、ニワトリ、カイコ)および栽培種2種(イネおよびダイズ)と、それぞれの野生種の全ゲノムリシークエンシングデータを用いた比較解析を行い、家畜化によるゲノム中への有害変異蓄積を調査しました。我々は、家畜種・栽培種において、野生種よりも遺伝的変異が有意に減少し、有害変異の指標となる同義置換に対する非同義置換の割合が増加していることを見出しました (Makino T. et al., GBE 2018)。こうした相違は、どのようなアリル頻度をもつ座位においても観察されました。また、脊椎動物のゲノムにおいて高度に保存された領域中において、家畜種の方が野生種よりもSNPの出現頻度が高い傾向を見出し、有害と分類される遺伝的変化の割合が家畜種で高い傾向を示しました。これらの結果は、自然選択の効率の低下をもたらす集団ボトルネックに起因すると考えられます。ここで得られた知見を元に家畜化・栽培化過程において影響力の大きな遺伝的変化を探索し、畜産・農業への応用を目指して研究を進めています。
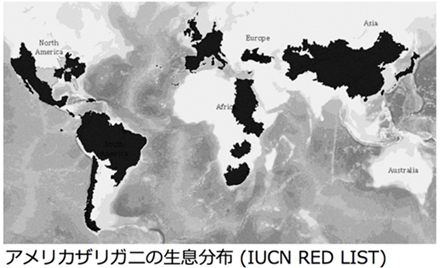
侵略的外来種アメリカザリガニのゲノム解析
世界各地で外来種が生態系を破壊する問題が深刻化していますが、他の地域から持ち込まれた生物種の全てが新規環境に定着しているわけではありません。特定の種が世界各地で独立に何度も侵略的外来種となっている背景には、これらの種に共通する遺伝的な基盤があると考えられますが、その詳細は分かっていません。北米原産のアメリカザリガニは、赤道地域から高緯度地域に至るまで世界中の様々な環境に適応し、現在も生息範囲を広げています。近年、高温水性である本種が寒冷地においても定着確認されるようになり、侵略的外来種の中にでも環境適応能力の高さが際立っています。私たちは、アメリカザリガニを対象に環境適応能力の高さを生み出す遺伝的多様性の調査や、環境適応能力の遺伝的基盤の解明を目指して研究を行なっています。